北大雷晓光团队揭示植物来源分子间Diels-Alder反应酶的自然演化奥秘
近日,北京大学化学与分子工程学院雷晓光(点击查看介绍)课题组在植物天然产物生物合成酶的演化机制研究中取得重要进展,深度解析了白桑中分子间Diels-Alder反应酶的演化历程。该工作发表在Nature Communications 上。
Diels-Alder(D-A)反应是有机合成中最重要的反应之一,广泛应用于天然产物的全合成之中。因此,从自然界中挖掘可以催化D-A反应的酶(D-A酶)是生物合成领域中的研究热点。在目前已知的D-A酶中,不同功能的D-A酶在序列和结构上缺乏明显的保守性,这一特征也使得新颖D-A酶的鉴定,特别是植物来源的D-A酶,变得更加困难。因此,对D-A酶演化机制的深入研究,有望揭示自然界“生成”这类酶的奥秘所在。这将使我们更深入地理解D-A酶产生的先决条件和后天影响因素,从而为D-A酶的理性挖掘和酶学改造提供新的视角和指引。
在实验室的前期研究中,雷晓光课题组发现了桑树中存在着两种不同endo/exo选择性的分子间D-A酶,并对其催化机制进行了详细研究(Nat. Chem. 2020, 12, 620–628,点击阅读详细; Nat. Catal. 2021, 4, 1059–1069,点击阅读详细)。在这些研究的基础上,作者发现桑科植物来源的分子间D-A酶MaDA与二烯体合成酶MaMO在序列和结构上呈现显著相似性,均隶属于FAD偶联氧化酶家族中的类BBE酶亚家族。值得注意的是,D-A酶催化的是非氧化反应,而其它类BBE家族的酶多催化氧化反应,这暗示MaDA酶很可能是该家族中新功能化的成员,这点引发了研究者的浓厚兴趣。值得一提的是,多种D-A酶如MaDA、Sol5和PyrE3被推测起源于FAD依赖的氧化酶,而MaDA正是这一代表性类别中的关键成员。深入研究D-A酶MaDA是如何从FAD偶联氧化酶中演化而来的,将有助于我们更全面地理解D-A酶的演化起源和机制。因此,作者选择以白桑来源的D-A酶为研究对象,旨在深入剖析其演化起源和机制,为揭示植物中这类关键酶的演化路径和功能演化提供有力的实证支持。
作者对D-A酶MaDA和二烯体合成酶MaMO的二级结构进行了深入分析,初步推测它们隶属于类BBE酶家族。为进一步明晰这两种酶与类BBE家族中其他酶的系统演化关系,作者系统收集了NCBI、Uniprot及PDB等权威公开数据库中关于类BBE家族酶的数据。鉴于当前公开数据库对白桑蛋白的注释尚显不足,作者对白桑转录组的原始下机测序数据进行了全面而细致的组装与注释工作。鉴于白桑属于桑科物种,为全面而深入地挖掘D-A酶MaDA的同源基因,作者还进一步对桑科其他物种的转录组数据进行了de novo组装和注释,并成功从桑科物种的蛋白组中鉴定出了类BBE家族的酶。

图1. 类BBE家族酶的系统发育树
经过上述严格的数据挖掘和深入分析,作者最终获取了8699个非冗余的类BBE家族酶序列。在此基础上,作者利用这些酶的数据,构建了一个更为精准和完整的非冗余类BBE家族酶系统发育树。通过系统发育树分析,作者发现D-A酶隶属于类BBE家族蛋白中5d亚家族,并形成了一个新功能化的分支(图1)。
随后作者进一步在桑科植物中挖掘到了更多D-A酶,并发现其独特地存在于桑科物种之中。为了揭示D-A酶的新功能来源,作者构建了包含D-A酶及其演化关系最紧密的酶在内的系统发育树,并将其划分为三个分支:Clade I、Clade II和Clade III(图2)。经过酶学实验,作者发现Clade I为氧化环化酶(OCs),Clade II为D-A酶(DAs),而Clade III为二烯体合成酶(DSs)。此外,作者还发现D-A酶和二烯体合成酶在酶活性上均展现出氧化环化酶的活性,但彼此之间不存在交叉活性。
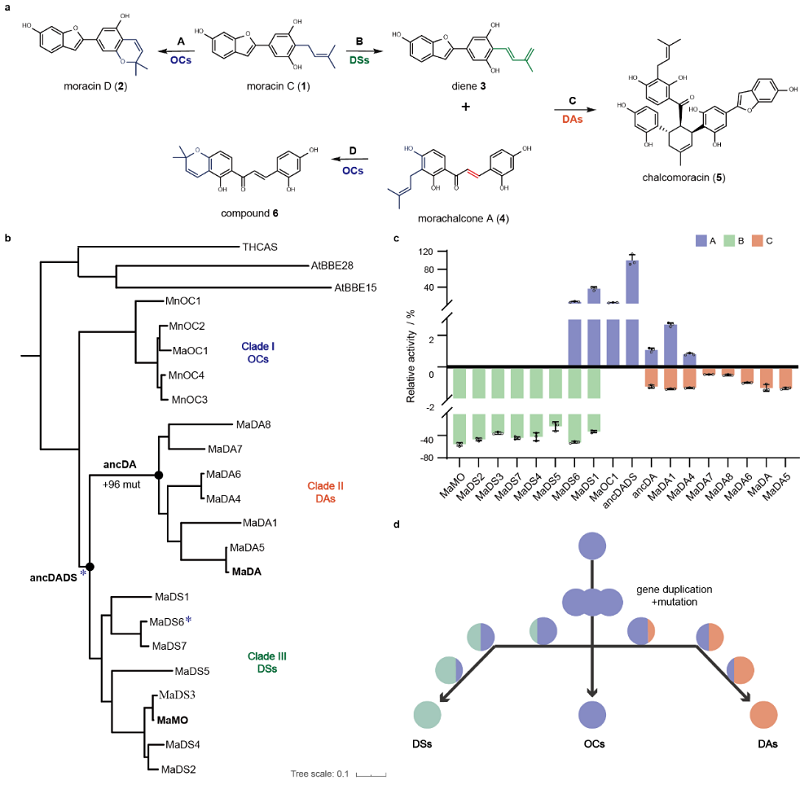
图2. D-A酶、二烯体合成酶和氧化环化酶的功能鉴定和演化模型
随后,作者通过祖先序列重构技术,构建了D-A酶和二烯体合成酶的共同祖先ancDADS,以及D-A酶的祖先ancDA。实验验证结果表明,ancDADS具有氧化环化酶活性,而ancDA则表现出双功能的D-A酶活性。也就是说,D-A酶和二烯体合成酶起源于氧化环化酶,且D-A酶的祖先泛杂着氧化环化酶的活性。基于对D-A酶、二烯体合成酶和氧化环化酶酶活性的深入比较与分析,作者推断出D-A酶的演化轨迹:从一个具备氧化活性的祖先酶出发,经过基因复制形成多个拷贝,在突变积累的过程中,酶获得了泛杂活性。随后,这些酶一方面演化为了二烯体合成酶,另一方面则演化为了D-A酶。
上述发现解答了D-A酶从何而来这一关键问题。然而作者的探索并未止步于此。作者进一步期望通过深入研究来解答氧化环化酶是如何演化为D-A酶这一关键科学问题。为此,作者全面地比较了氧化环化酶与D-A酶这两类酶的结构差异,对MaDA1、ancDA和ancDADS三种酶的结构进行了细致地叠加对比分析,特别关注了它们活性口袋区域的异同(图3)。通过对比分析,作者发现这三种酶均遵循典型的类BBE酶家族的结构特征。它们的活性口袋均由六个β折叠(β12-14,β16-β18)、一个α螺旋(α11)以及一个loop环构成,并且都含有辅因子FAD。作者随后发现氧化环化酶和D-A酶的酶活性口袋处总共有21个差异残基(图3d)。
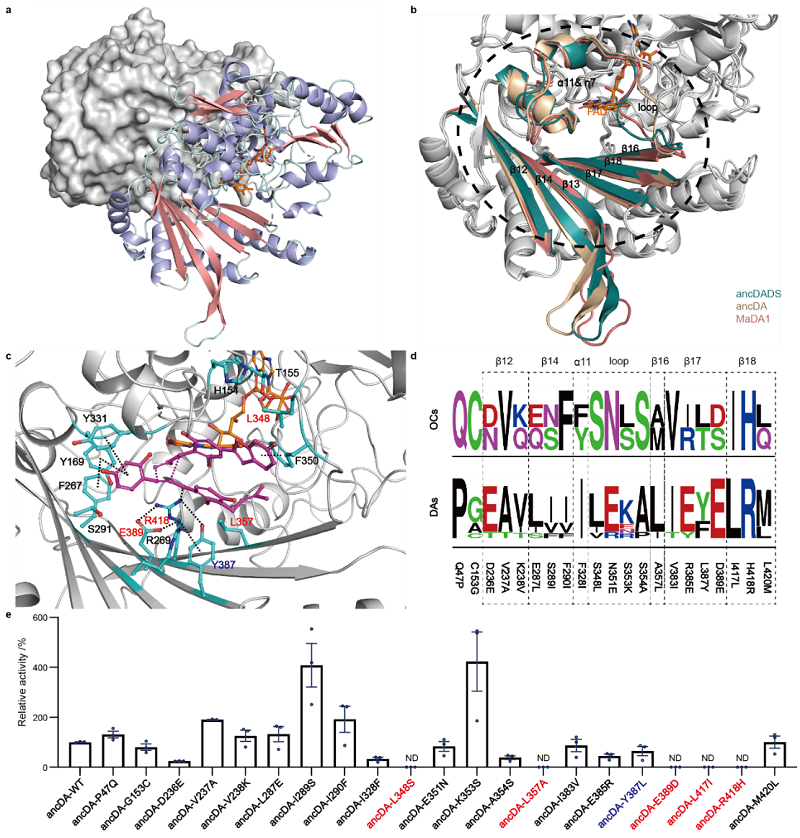
图3. D-A酶的结构、活性口袋和关键突变残基
随后,作者对ancDA和底物过渡态进行了分子动力学模拟的计算。作者发现ancDA中E389可以与R418形成氢键,而R418可以与亲二烯体形成氢键相互作用,从而催化D-A反应的发生,这一发现与课题组前期对于MaDA的催化机制研究结论相同。除此之外,作者还发现Y387这一差异残基可以和R418发生氢键相互作用,间接参与了亲二烯体4的活化。
为了深入探究这21个残基对D-A酶活性的影响,作者将ancDADS中相应的残基突变到ancDA中,构建了21个带有不同单点突变位点的ancDA突变体,如ancDA-P47Q、ancDA-G153C等,并对这些突变体进行了酶活性测试。结果发现,在21个突变体中,有5个突变体(ancDA-L348S、ancDA-L357A、ancDA-E389D、ancDA-L417I和ancDA-R418H)不再具备催化Diels-Alder反应的活性(图3e)。这一发现表明这五个位点的氨基酸替换在氧化环化酶演化出D-A酶活性的过程中起着关键作用。
为了进一步验证这五个残基在从氧化环化酶演化为D-A酶过程中的决定性作用,作者构建了包含这五个突变的ancDADS的突变体,命名为ancDADS-S348L-A357L-D389E-I417L-H418R(简称ancDADS-mut5a)。随后,作者进行了ancDADS-mut5a的分子克隆和蛋白质表达,并通过酶学实验详细评估了其酶活性,结果发现ancDADS-mut5a具备D-A酶的活性(图4)。在这五个关键残基中,E389和R418扮演着催化残基的角色,而L348和L357则作为辅助结合残基。相比之下,I417L位于酶的活性口袋的背面,不与底物发生直接相互作用。因此,作者推测该位点对于酶的D-A活性演化可能不是决定性因素。为了验证这一假设,作者构建了移除了I417L突变的ancDADS-mut4。通过活性测试,作者发现尽管ancDADS-mut4的D-A酶活性较弱,但仍然具有一定的催化能力(图4)。然而,删除这四个突变中的任何一个或者两个,ancDADS-mut4的D-A酶活性基本消失,这证明了这四个突变对于D-A酶的活性出现是充分且必要的(图4)。

图4. 氧化环化酶获得D-A酶活性的关键组合突变
作者还探索了L387Y和V156I对酶演化的作用,作者发现带有L387Y的ancDADS-mut5a的D-A酶活性进一步增强,而携带V156I突变的组合突变体的氧化环化活性降低。这表明在氧化环化酶演化为D-A酶的过程中,除了关键的几个残基参与到Diels-Alder活性的出现之外,还有其它的残基参与到Diels-Alder活性的增强以及原始氧化环化活性的减弱(图4)。
在接下来的深入研究中,作者致力于探索氧化环化酶向D-A酶演化过程中底物结合模式的变化。通过细致比较moracin C和亲二烯体4在ancDADS中的结合模式,作者发现这两种底物在酶口袋中占据了高度重叠的空间位置(图5a)。这种底物的叠加模式强烈暗示了它们可能以竞争性的方式与ancDADS结合。为了验证这一假设,作者进行了酶的抑制常数Ki的测定实验。在实验过程中,作者向ancDADS和moracin C的反应体系中逐渐加入了不同浓度的亲二烯体4,并实时监测了ancDADS对moracin C的催化速度变化。实验数据表明,随着亲二烯体4浓度的递增,ancDADS催化Moracin C生成产物的速率呈现显著下降趋势。通过精确计算,作者确定了亲二烯体4对ancDADS催化moracin C这一反应的抑制常数Ki为7.343 μM,同时观察到最大反应速度(Vmax)保持恒定(图5b)。这些实验结果强烈支持了上述的假设,即亲二烯体4和moracin C是竞争性结合在ancDADS中的。
随后,作者对ancDA、ancDADS-mut4、ancDADS以及MaDA1的FAD与底物过渡态之间反应中心的距离进行了精确的计算,并将相关的径向分布函数以图表的形式展示于图5c中。研究结果表明,MaDA1、ancDA以及ancDADS-mut4的底物过渡态距离大约为4 Å,而ancDADS与底物反应中心的距离则显著增加到大约14Å。这一发现具有重要意义,因为它暗示了这五个残基的突变不仅使氧化环化酶从不能容纳两个底物,转变为能够同时容纳两个底物,而且还能维持底物在D-A酶中的特定构象,从而确保其催化活性。
基于以上实验,作者提出了D-A酶的演化机制(图5e)。D-A酶是从同家族的氧化环化酶演化而来,该祖先氧化环化酶具有结合两个底物——moracin C和亲二烯体4——的能力,并分别催化它们进行氧化环化反应。通过基因复制过程,祖先酶产生了多个拷贝,这些拷贝在突变和自然选择的共同作用下,逐步获得了新的功能特性。其中一部分酶演化为了二烯体合成酶,该酶能够高效地催化moracin C氧化生成二烯体3,从而为D-A酶提供了关键底物。另一方面,另一部分酶则经历了一系列关键突变,降低了原有的氧化环化活性,同时通过S348L、A357L、L387Y、D389E、I417L和H418R等替换,获得了催化Diels-Alder反应的活性,最终演化成为单功能D-A酶。
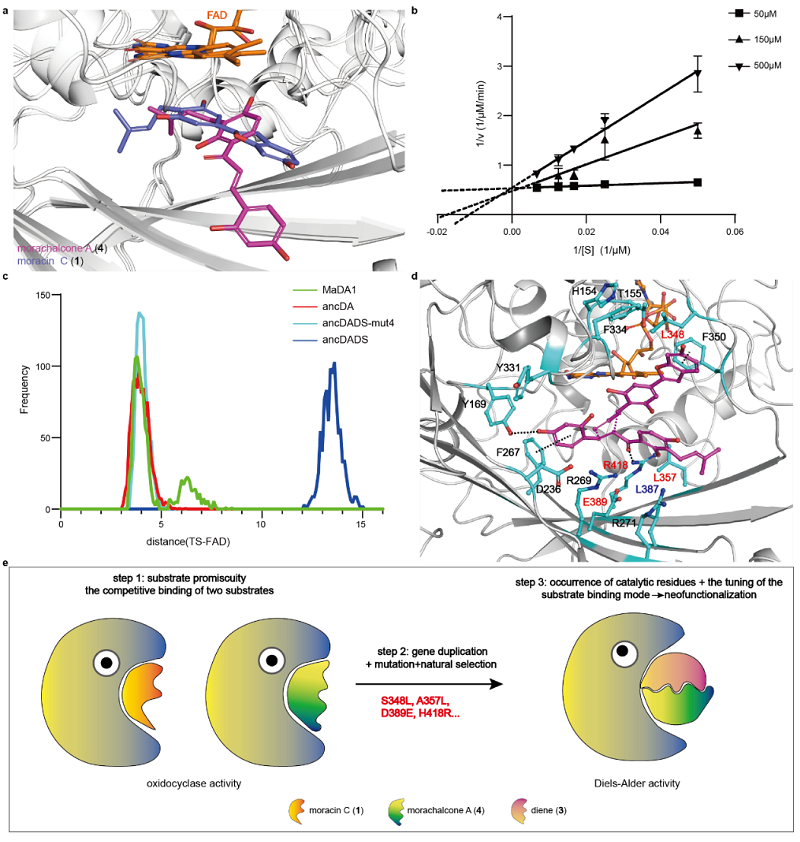
图5. 演化过程中底物结合模式的改变
综上所述,作者综合运用了系统发育树构建、祖先序列重建、蛋白表达和酶活性检测等多种方法,系统揭示了桑树中D-A酶的演化历程。在这一过程中,氧化环化酶经过基因复制事件,形成了D-A酶及其功能相关的二烯体合成酶的共同祖先。为了阐明FAD依赖的氧化环化酶向D-A酶演化的具体机制,作者进一步开展了晶体结构分析、分子动力学模拟以及定点突变实验。研究结果显示,在演化过程中,关键氨基酸残基的替换(如A357L、S348L、D389E和H418R等)赋予了酶新的催化分子间Diels-Alder反应的能力。此外,突变如I417L和L387Y显著增强了Diels-Alder活性,而I156V等突变则有效降低了原有的氧化环化活性,推动了氧化环化酶向单功能D-A酶的功能转变。作者还发现酶底物的微调在演化过程中起到了至关重要的作用。原本只能竞争性容纳单一底物的酶,通过演化能够容纳两个底物以进行D-A反应。该研究不仅深化了我们对D-A酶形成机制的理解,也加深了我们对植物天然产物结构多样性的酶学机制的认识,更重要的是,它为新颖D-A酶的理性挖掘和改造提供了重要的理论指导,对于D-A酶的进一步开发和利用具有重要科学意义。
北京大学化学与分子工程学院、北大-清华生命科学联合中心雷晓光教授和团队中高磊特聘副研究员为论文的共同通讯作者。北京大学雷晓光课题组博士生丁琪和郭念昕为共同第一作者;范俊萍特聘副研究员、吴东山、杨军以及美国东北大学翁经科教授和其研究生Michelle McKee也参与了相关研究工作。相关生物信息学分析和计算化学在北京大学高性能计算平台上完成。该研究得到了国家重点研发计划、国家自然科学基金委员会、北京分子科学国家研究中心、北大-清华生命科学联合研究中心、新基石基金会等项目和单位的资助。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

The evolutionary origin of naturally occurring intermolecular Diels-Alderases from Morus alba
Qi Ding, Nianxin Guo, Lei Gao, Michelle McKee, Dongshan Wu, Jun Yang, Junping Fan, Jing-Ke Weng & Xiaoguang Lei
Nat. Commun., 2024, 15, 2492, DOI: 10.1038/s41467-024-46845-0
导师介绍
雷晓光
https://www.x-mol.com/groups/lei_xiaoguang
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!




























 京公网安备 11010802027423号
京公网安备 11010802027423号