Journal of Web Semantics ( IF 2.5 ) Pub Date : 2022-09-14 , DOI: 10.1016/j.websem.2022.100756 Nikola Milošević , Wolfgang Thielemann
|
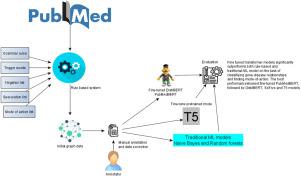
Biomedical research is growing at such an exponential pace that scientists, researchers, and practitioners are no more able to cope with the amount of published literature in the domain. The knowledge presented in the literature needs to be systematized in such a way that claims and hypotheses can be easily found, accessed, and validated. Knowledge graphs can provide such a framework for semantic knowledge representation from literature. However, in order to build a knowledge graph, it is necessary to extract knowledge as relationships between biomedical entities and normalize both entities and relationship types. In this paper, we present and compare a few rule-based and machine learning-based (Naive Bayes, Random Forests as examples of traditional machine learning methods and DistilBERT, PubMedBERT, T5, and SciFive-based models as examples of modern deep learning transformers) methods for scalable relationship extraction from biomedical literature, and for the integration into the knowledge graphs. We examine how resilient are these various methods to unbalanced and fairly small datasets. Our experiments show that transformer-based models handle well both small (due to pre-training on a large dataset) and unbalanced datasets. The best performing model was the PubMedBERT-based model fine-tuned on balanced data, with a reported F1-score of 0.92. The distilBERT-based model followed with an F1-score of 0.89, performing faster and with lower resource requirements. BERT-based models performed better than T5-based generative models.
中文翻译:

用于知识图谱创建的生物医学关系提取方法和模型比较
生物医学研究正以指数级的速度增长,以至于科学家、研究人员和从业者不再能够应对该领域发表的大量文献。文献中呈现的知识需要以这样的方式系统化,以便可以轻松找到、访问和验证主张和假设。知识图谱可以为文献中的语义知识表示提供这样的框架。然而,为了构建知识图谱,有必要将知识提取为生物医学实体之间的关系,并对实体和关系类型进行规范化。在本文中,我们介绍并比较了一些基于规则和基于机器学习(朴素贝叶斯、随机森林作为传统机器学习方法的示例和 DistilBERT、PubMedBERT、T5、和基于 SciFive 的模型作为现代深度学习转换器的示例)用于从生物医学文献中提取可扩展关系以及集成到知识图谱中的方法。我们研究了这些不同方法对不平衡和相当小的数据集的弹性。我们的实验表明,基于转换器的模型可以很好地处理小型(由于对大型数据集进行预训练)和不平衡数据集。表现最好的模型是基于 PubMedBERT 的模型,它在平衡数据上进行了微调,报告的 F1 分数为 0.92。基于 distilBERT 的模型的 F1 分数为 0.89,性能更快且资源需求更低。基于 BERT 的模型比基于 T5 的生成模型表现更好。以及集成到知识图谱中。我们研究了这些不同方法对不平衡和相当小的数据集的弹性。我们的实验表明,基于转换器的模型可以很好地处理小型(由于对大型数据集进行预训练)和不平衡数据集。表现最好的模型是基于 PubMedBERT 的模型,它在平衡数据上进行了微调,报告的 F1 分数为 0.92。基于 distilBERT 的模型的 F1 分数为 0.89,性能更快且资源需求更低。基于 BERT 的模型比基于 T5 的生成模型表现更好。以及集成到知识图谱中。我们研究了这些不同方法对不平衡和相当小的数据集的弹性。我们的实验表明,基于转换器的模型可以很好地处理小型(由于对大型数据集进行预训练)和不平衡数据集。表现最好的模型是基于 PubMedBERT 的模型,它在平衡数据上进行了微调,报告的 F1 分数为 0.92。基于 distilBERT 的模型的 F1 分数为 0.89,性能更快且资源需求更低。基于 BERT 的模型比基于 T5 的生成模型表现更好。表现最好的模型是基于 PubMedBERT 的模型,它在平衡数据上进行了微调,报告的 F1 分数为 0.92。基于 distilBERT 的模型的 F1 分数为 0.89,性能更快且资源需求更低。基于 BERT 的模型比基于 T5 的生成模型表现更好。表现最好的模型是基于 PubMedBERT 的模型,它在平衡数据上进行了微调,报告的 F1 分数为 0.92。基于 distilBERT 的模型的 F1 分数为 0.89,性能更快且资源需求更低。基于 BERT 的模型比基于 T5 的生成模型表现更好。



























 京公网安备 11010802027423号
京公网安备 11010802027423号